Семинар¶
Теорема Гаусса-Маркова.¶
Теорема. Если
\(y = X\beta + u\), \(\beta\) – константы.
Оценивается регрессия \(\hat{y} = X\hat{\beta}\) при помощи МНК.
Матрица \(X\) детерминирована и имеет полный ранг.
\(\mathbb{E}(u) = 0, \mathrm{Var}(u) = \sigma^2 I\).
\(n >> k\),
то
\(\hat{\beta}\) существуют и единственны.
\(\hat{\beta}\) линейны по \(y\).
\(\mathbb{E}(\hat{\beta}) = \beta\).
\(\hat{\beta}\) эффективны в классе линейных по \(y\) несмещённых оценок.
Гетероскедастичность.¶
Определение.¶
Нарушение предпосылки ТГМ: \(\mathrm{Var}(u_i) = \sigma_i^2 \ne \sigma\).
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
sns.set()
# Статистика
import statsmodels.formula.api as smf # Модели и гипотезы
import statsmodels.stats.api as sms # Тесты
x = np.arange(50)
u = [np.random.normal(0, i) for i in range(50)]
y = 3 + 2 * x + u
plt.scatter(x, y)
plt.plot(x, y - u, c='r')
plt.xlabel('x')
plt.ylabel('y')
plt.title('Гетероскедастичность')
Text(0.5, 1.0, 'Гетероскедастичность')
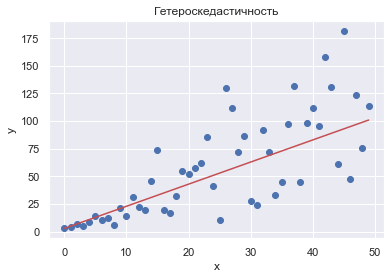
Проблема: \(\hat{\sigma}^2 = \dfrac{RSS}{n-k}\) неверна, а значит неверны \(\mathrm{se}(\hat{\beta})\), а значит неверно строятся доверительные интервалы и нельзя доверять результатам проверки гипотез.
Идентификация.¶
Существует большое количество различных статистических тестов. Например:
Goldfeld-Quandt test.
Тестируется гипотеза \(H_0\) против \(H_1\): $\( \begin{cases} H_0: \sigma_i^2 = \sigma^2, \\ H_1: \sigma_i \sim X_j. \end{cases} \)$
Схема:
Отсортировать регрессор \(X_j\) по возрастанию.
Разбить отсортированный ряд на три одинаковые части. Предполагается, что первая часть имеет случайную ошибку \(\sigma^2_1\), а третья часть \(\sigma^2_2\). Вторая часть выбрасывается.
Проверить гипотезу $\( \begin{cases} H_0: \sigma_1^2 = \sigma_2^2, \\ H_1: \sigma_1^2 \ne \sigma_2^2. \end{cases} \)$
Для этого оцениваем модель по первой части наблюдений и получаем \(RSS_1\), оцениваем по второй части наблюдений и получаем \(RSS_2\), а затем используем \(F\)-статистику:
Если \(H_0\) отвергается, то в данных наблюдается гетероскедастичность из-за \(X_j\).
data.shape[0] / 3 - 6 # F_{17974, 17974}
17974.0
data = sns.load_dataset('diamonds')
data.head()
| carat | cut | color | clarity | depth | table | price | x | y | z | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0.23 | Ideal | E | SI2 | 61.5 | 55.0 | 326 | 3.95 | 3.98 | 2.43 |
| 1 | 0.21 | Premium | E | SI1 | 59.8 | 61.0 | 326 | 3.89 | 3.84 | 2.31 |
| 2 | 0.23 | Good | E | VS1 | 56.9 | 65.0 | 327 | 4.05 | 4.07 | 2.31 |
| 3 | 0.29 | Premium | I | VS2 | 62.4 | 58.0 | 334 | 4.20 | 4.23 | 2.63 |
| 4 | 0.31 | Good | J | SI2 | 63.3 | 58.0 | 335 | 4.34 | 4.35 | 2.75 |
model = smf.ols('np.log(price) ~ depth + table + x + y + z', data = data).fit()
model.summary()
| Dep. Variable: | np.log(price) | R-squared: | 0.919 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.919 |
| Method: | Least Squares | F-statistic: | 1.226e+05 |
| Date: | Thu, 12 Nov 2020 | Prob (F-statistic): | 0.00 |
| Time: | 00:07:49 | Log-Likelihood: | -9493.6 |
| No. Observations: | 53940 | AIC: | 1.900e+04 |
| Df Residuals: | 53934 | BIC: | 1.905e+04 |
| Df Model: | 5 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| Intercept | 2.8430 | 0.079 | 35.811 | 0.000 | 2.687 | 2.999 |
| depth | 0.0093 | 0.001 | 8.986 | 0.000 | 0.007 | 0.011 |
| table | -0.0109 | 0.001 | -18.413 | 0.000 | -0.012 | -0.010 |
| x | 0.7973 | 0.007 | 118.034 | 0.000 | 0.784 | 0.811 |
| y | 0.0365 | 0.005 | 7.425 | 0.000 | 0.027 | 0.046 |
| z | 0.0615 | 0.009 | 7.204 | 0.000 | 0.045 | 0.078 |
| Omnibus: | 24199.104 | Durbin-Watson: | 1.346 |
|---|---|---|---|
| Prob(Omnibus): | 0.000 | Jarque-Bera (JB): | 2130668.668 |
| Skew: | 1.252 | Prob(JB): | 0.00 |
| Kurtosis: | 33.688 | Cond. No. | 5.43e+03 |
Warnings:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
[2] The condition number is large, 5.43e+03. This might indicate that there are
strong multicollinearity or other numerical problems.
?sms.het_goldfeldquandt
# GQ-test: the first value is F-stat
sms.het_goldfeldquandt(model.model.endog, model.model.exog)
# H0 не отвергается, гетероскедастичность не выявлена
(0.5571425675792742, 0.9999999999999999, 'increasing')
Breush-Pagan test.
Тестируется гипотеза \(H_0\) против \(H_1\):
где \(f\) – некоторая гладкая функция, \(Z\) – некоторые регрессоры, которые могут быть и не включены в нашу модель.
Схема:
Оценить регрессию \(\hat{y} = X\hat{\beta}\).
Получить оценку \(\hat{\sigma}^2 = \dfrac{RSS}{n}\) (смещённая).
Создать новую переменную \(q := \dfrac{y-\hat{y}}{\hat{\sigma}^2}\) (поэлементно).
Оценить регрессию \(\hat{q} = Z\hat{\alpha}\).
Протестировать \(H_0\) при помощи статистики $\( \chi^2 = \dfrac{ESS}{2} \sim \chi^2_{\text{число регрессоров в \)Z\(, не считая константный}}. \)$
?sms.het_breuschpagan
# Первое значение -- значение статистики
sms.het_breuschpagan(model.resid, model.model.exog)
# H0 отвергается, гетероскедастичность выявлена
(318.88832638372446,
8.68231926481354e-67,
64.14981882459652,
5.505510514064581e-67)
White test.
Тестируется гипотеза \(H_0\) против \(H_1\):
Схема:
Оценить регрессию \(\hat{y} = X\hat{\beta}\).
Оценить регрессию \((y_i - \hat{y}_i)^2 = a_0 + \sum_jb_jX_j + \sum_jc_jX_j^2 + \sum_{i<j}d_jX_iX_j\). Найти \(R^2\) в этой регрессии.
Проверить \(H_0\) при помощи статистики \(\chi^2 = n \times R^2 \sim \chi^2_{1 + j + j + (j^2 - j)/2}\).
?sms.het_white
# Первое значение -- значение статистики
sms.het_white(model.resid, model.model.exog)
# H0 отвергается, гетероскедастичность выявлена
(8939.302502210943, 0.0, 535.5453119814345, 0.0)
Корректировка гетероскедастичности.¶
I. В теории: пусть знаем \(\sigma_i^2\) для всех \(i\). В этом случае мы можем “исправить” модель так, чтобы гетероскедастичности не было.
Задание 1. Рассмотрим модель \(y_i = \beta_0 + \beta_1x_i + u_i\), \(\mathrm{Var}(u_i) = \dfrac{\sigma^2}{(3 + x_i)^2}\). Скорректируйте гетероскедастичность в модели и выпишите выражение для оценок скорректированной модели.
Решение. Заметим, что дисперсия случайной ошибки представляется в виде \(\mathrm{Var}(u_i) = \sigma^2 \times f(x_i).\) Поделим каждый компонент регрессии на \(\sqrt{f(x_i)}\):
Переобозначим: \((3 + x_i)y_i =: \tilde{y}_i\), \(\beta_0(3+x_i) =: \beta_0\tilde{X}_{1i}\), \(\beta_1x_i(3+x_i) =: \beta_1\tilde{X}_{2i}\), \((3+x_i)u_i =: \tilde{u}_i\). Заметим, что в модели
гетероскедастичности нет, потому что
а значит, к ней применима ТГМ. Оценки в этой модели можно рассчитать по стандартной формуле:
Задание 2. Рассмотрим модель \(y = X\beta + u\), \(\mathrm{Var}(u) = \sigma^2A\), \(A\) известна.
Скорректируйте гетероскедастичность в модели и выпишите выражение для оценок этой модели.
Выведите дисперсию оценок скорректированной модели.
Выведите дисперсию оценок МНК.
Решение.
Как и в предыдущей задаче, корректировка производится делением компонентов модели на корень из функции, стоящей при \(\sigma\). В матричном случае эта операция превращается в домножение на \(A^{-1/2}\), где
где \(P\) – матрица собственных векторов.
Скорректированная модель:
Заметим, что
II. На практике: \(\sigma_i^2\) не знаем. Тогда есть три варианта.
Вариант 1: исправить только доверительные инетрвалы для параметров модели, оцениваемой при помощи МНК,так, чтобы они были устойчивы к гетероскедастичности. Тогда
\(\hat{\mathrm{Var}}(u)_{HC}\) можно взять разными способами:
\(HC_0\): \(\hat{\mathrm{Var}}(u)_{HC_0} = \mathrm{diag}(\hat{u}^2_1, \ldots, \hat{u}^2_n)\).
\(HC_3\): \(\hat{\mathrm{Var}}(u)_{HC_3} = \mathrm{diag}(\hat{u}^2_{1, CV}, \ldots, \hat{u}^2_{n, CV})\).
Задание 3. Рассмотрим модель \(y_i = \beta x_i + u_i\). Известно, что \(y_1 = y_2 = y_3 = 2\), \(x_1 = x_2 = 1\), \(x_3 = 4\).
Найдите \(\hat{\beta}_{OLS}\).
Найдите \(\hat{\mathrm{Var}}(\hat{\beta_{OLS}})_{HC_0}\).
Найдите \(\hat{\mathrm{Var}}(\hat{\beta_{OLS}})_{HC_3}\).
Решение.
Вспомним, что в модели парной регрессии без константы МНК-оценка коэффициента строится следующим образом:
Найдём остатки:
Тогда
Чтобы получить остатки на кросс-валидации, нужно для \(i\)-го наблюдения оценить модель на прочих \(n-1\) наблюдениях и рассчитать остатки по полученным оценкам. Для нашего примера:
\(y_i\) |
\(x_i\) |
\(\hat{\beta}_{CV}\) |
\(\hat{y}_{CV}\) |
\(\hat{u}_{CV}\) |
|---|---|---|---|---|
2 |
1 |
\(\frac{10}{17}\) |
\(\frac{10}{17}\) |
\(\frac{24}{17}\) |
2 |
1 |
\(\frac{10}{17}\) |
\(\frac{10}{17}\) |
\(\frac{24}{17}\) |
2 |
4 |
2 |
8 |
-6 |
Тогда
model.cov_params()
| Intercept | depth | table | x | y | z | |
|---|---|---|---|---|---|---|
| Intercept | 0.006303 | -7.520541e-05 | -2.917492e-05 | -1.201055e-04 | -3.607090e-05 | 2.583795e-04 |
| depth | -0.000075 | 1.077368e-06 | 1.561260e-07 | 2.118084e-06 | 4.742715e-07 | -4.281410e-06 |
| table | -0.000029 | 1.561260e-07 | 3.530415e-07 | -2.831128e-07 | 1.047273e-07 | 7.671272e-08 |
| x | -0.000120 | 2.118084e-06 | -2.831128e-07 | 4.562656e-05 | -2.053421e-05 | -3.904137e-05 |
| y | -0.000036 | 4.742715e-07 | 1.047273e-07 | -2.053421e-05 | 2.417944e-05 | -5.709749e-06 |
| z | 0.000258 | -4.281410e-06 | 7.671272e-08 | -3.904137e-05 | -5.709749e-06 | 7.293067e-05 |
model.cov_HC0
array([[ 2.53646463e-02, -3.79811138e-04, -3.70015534e-05,
-1.64932777e-03, -1.37831330e-03, 4.96374117e-03],
[-3.79811138e-04, 6.02318499e-06, 1.90578318e-07,
3.32326293e-05, 1.65964402e-05, -8.15647500e-05],
[-3.70015534e-05, 1.90578318e-07, 4.89255718e-07,
-6.41399781e-06, 5.51885356e-06, 6.46117971e-07],
[-1.64932777e-03, 3.32326293e-05, -6.41399781e-06,
1.70474508e-03, -1.30321102e-03, -6.59553761e-04],
[-1.37831330e-03, 1.65964402e-05, 5.51885356e-06,
-1.30321102e-03, 1.42545309e-03, -1.88329799e-04],
[ 4.96374117e-03, -8.15647500e-05, 6.46117971e-07,
-6.59553761e-04, -1.88329799e-04, 1.38362462e-03]])
model.HC0_se
Intercept 0.159263
depth 0.002454
table 0.000699
x 0.041289
y 0.037755
z 0.037197
dtype: float64
# Модель можно переоценить с учётом робастных стандартных ошибок
model_hc = smf.ols('np.log(price) ~ depth + table + x + y + z', data = data).fit(cov_type = 'HC0')
model_hc.summary()
| Dep. Variable: | np.log(price) | R-squared: | 0.919 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.919 |
| Method: | Least Squares | F-statistic: | 4.141e+04 |
| Date: | Thu, 12 Nov 2020 | Prob (F-statistic): | 0.00 |
| Time: | 00:37:09 | Log-Likelihood: | -9493.6 |
| No. Observations: | 53940 | AIC: | 1.900e+04 |
| Df Residuals: | 53934 | BIC: | 1.905e+04 |
| Df Model: | 5 | ||
| Covariance Type: | HC0 |
| coef | std err | z | P>|z| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| Intercept | 2.8430 | 0.159 | 17.851 | 0.000 | 2.531 | 3.155 |
| depth | 0.0093 | 0.002 | 3.800 | 0.000 | 0.005 | 0.014 |
| table | -0.0109 | 0.001 | -15.642 | 0.000 | -0.012 | -0.010 |
| x | 0.7973 | 0.041 | 19.310 | 0.000 | 0.716 | 0.878 |
| y | 0.0365 | 0.038 | 0.967 | 0.334 | -0.037 | 0.111 |
| z | 0.0615 | 0.037 | 1.654 | 0.098 | -0.011 | 0.134 |
| Omnibus: | 24199.104 | Durbin-Watson: | 1.346 |
|---|---|---|---|
| Prob(Omnibus): | 0.000 | Jarque-Bera (JB): | 2130668.668 |
| Skew: | 1.252 | Prob(JB): | 0.00 |
| Kurtosis: | 33.688 | Cond. No. | 5.43e+03 |
Warnings:
[1] Standard Errors are heteroscedasticity robust (HC0)
[2] The condition number is large, 5.43e+03. This might indicate that there are
strong multicollinearity or other numerical problems.
Вариант 2. Использовать FGLS.
\(\hat{\Omega}\) можно оценивать по-разному: Пример 1, Пример 2, Пример 3, Пример 4. Можно использовать итерационный алгоритм, описанный, например, в Википедии.
Метод полезен, когда у нас есть некоторое представление о том, как устроена ковариационная матрица случайных ошибок.
from statsmodels.regression.linear_model import OLS, GLS
# Например, как описано в примере 3
log_resid = np.log(model.resid ** 2)
hat_omega_model = OLS(log_resid, model.model.exog).fit()
weights = 1 / np.exp(model.model.exog @ hat_omega_model.params)
weights
array([71.38203285, 90.7696654 , 99.93012519, ..., 59.07510695,
49.6225778 , 47.19220893])
model_gls = GLS(model.model.endog, model.model.exog, sigma = weights).fit()
model_gls.summary()
| Dep. Variable: | y | R-squared: | 0.918 |
|---|---|---|---|
| Model: | GLS | Adj. R-squared: | 0.918 |
| Method: | Least Squares | F-statistic: | 1.212e+05 |
| Date: | Thu, 12 Nov 2020 | Prob (F-statistic): | 0.00 |
| Time: | 00:43:41 | Log-Likelihood: | -11563. |
| No. Observations: | 53940 | AIC: | 2.314e+04 |
| Df Residuals: | 53934 | BIC: | 2.319e+04 |
| Df Model: | 5 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| const | 2.9610 | 0.075 | 39.314 | 0.000 | 2.813 | 3.109 |
| x1 | 0.0104 | 0.001 | 11.192 | 0.000 | 0.009 | 0.012 |
| x2 | -0.0118 | 0.001 | -18.941 | 0.000 | -0.013 | -0.011 |
| x3 | 0.8470 | 0.002 | 475.091 | 0.000 | 0.844 | 0.851 |
| x4 | -0.0076 | 0.000 | -25.813 | 0.000 | -0.008 | -0.007 |
| x5 | 0.0142 | 0.002 | 5.840 | 0.000 | 0.009 | 0.019 |
| Omnibus: | 7143.461 | Durbin-Watson: | 1.381 |
|---|---|---|---|
| Prob(Omnibus): | 0.000 | Jarque-Bera (JB): | 93702.657 |
| Skew: | -0.047 | Prob(JB): | 0.00 |
| Kurtosis: | 9.456 | Cond. No. | 5.05e+03 |
Warnings:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
[2] The condition number is large, 5.05e+03. This might indicate that there are
strong multicollinearity or other numerical problems.
III. Использовать бутстрэп.
Pairs Bootstrap.
Разные реализации, например, так.
from statsmodels.api import OLS
n_reps = 100
coef_matr = np.zeros((n_reps, 6))
for i in range(n_reps):
# 1. Строим бутстрэп-выборку
random_inds = np.random.randint(0, len(model.model.endog), size = len(model.model.endog))
y_b = model.model.endog[random_inds]
X_b = model.model.exog[random_inds, :]
# 2. Оцениваем модель и сохраняем оценки
model_b = OLS(y_b, X_b).fit()
coefs = np.array(model_b.params).reshape(1, -1)
coef_matr[i, :] = coefs
# Центрируем оценку каждого коэффициента
coef_matr = coef_matr - np.mean(coef_matr, axis = 0)
# Собираем ковариационную матрицу
cov_matr = 0
for i in range(n_reps):
cov_matr += coef_matr[i, :].reshape(-1, 1) @ coef_matr[i, :].reshape(-1, 1).T
pd.DataFrame(cov_matr / (n_reps - 1), columns = model.model.exog_names, index = model.model.exog_names)
| Intercept | depth | table | x | y | z | |
|---|---|---|---|---|---|---|
| Intercept | 0.099790 | -0.001459 | -0.000153 | 0.024355 | -0.037759 | 0.021478 |
| depth | -0.001459 | 0.000022 | 0.000001 | -0.000211 | 0.000421 | -0.000338 |
| table | -0.000153 | 0.000001 | 0.000001 | -0.000170 | 0.000176 | -0.000009 |
| x | 0.024355 | -0.000211 | -0.000170 | 0.042448 | -0.043042 | 0.000539 |
| y | -0.037759 | 0.000421 | 0.000176 | -0.043042 | 0.045780 | -0.003994 |
| z | 0.021478 | -0.000338 | -0.000009 | 0.000539 | -0.003994 | 0.005586 |
model.cov_params()
| Intercept | depth | table | x | y | z | |
|---|---|---|---|---|---|---|
| Intercept | 0.006303 | -7.520541e-05 | -2.917492e-05 | -1.201055e-04 | -3.607090e-05 | 2.583795e-04 |
| depth | -0.000075 | 1.077368e-06 | 1.561260e-07 | 2.118084e-06 | 4.742715e-07 | -4.281410e-06 |
| table | -0.000029 | 1.561260e-07 | 3.530415e-07 | -2.831128e-07 | 1.047273e-07 | 7.671272e-08 |
| x | -0.000120 | 2.118084e-06 | -2.831128e-07 | 4.562656e-05 | -2.053421e-05 | -3.904137e-05 |
| y | -0.000036 | 4.742715e-07 | 1.047273e-07 | -2.053421e-05 | 2.417944e-05 | -5.709749e-06 |
| z | 0.000258 | -4.281410e-06 | 7.671272e-08 | -3.904137e-05 | -5.709749e-06 | 7.293067e-05 |
Wild Bootstrap.
(Этот метод нужно будет реализовать в домашнем задании самостоятельно).
